分子モデリング・
シミュレーションソフトウェア
Discovery Studio
DS Protein Aggregation
タンパク質上の凝集しやすい部位の大きさと場所を特定
DS Protein Aggregationは、タンパク質上の凝集しやすい部位の大きさと場所を特定し、安定性の強化につながるmutationを予測します。マサチューセッツ工科大学のBernhaldt Trout教授が開発し、実験によって検証された“Spatial Aggregation Propensity(SAP)”アルゴリズムを利用しています。
抗体医薬などタンパク質その物を医薬として用いる場合、投与濃度が高いことから、その凝集が製剤設計上の大きな課題となっています。タンパク質の凝集は抗体に限らず、タンパク質の結晶化や創薬ターゲットタンパク質の調製(可溶化)においても、研究者を悩ます問題です。
アミノ酸の疎水性パラメータと表面への露出度から計算されるSAP値と、疎水性パッチ(表面)を見出すユニークなアルゴリズムにより、従来の類似の手法でははっきり確認することが難しかった(配列上は離れた残基から構成される)凝集面の予測はもちろん、凝集を回避するためにmutationを入れるべき残基の特定も、非常に簡単に行うことができます。アクセルリスではコードの最適化を行い、オリジナルに比較して100倍以上の高速化を達成しています。
DS Protein Aggregationによって変換すべきアミノ酸が特定できた場合、その変換によってタンパク質の安定性がどの程度変化するか、また変換位置がリガンドとの接触面に含まれる場合、リガンドとの親和性がどの程度変化するかを、Discovery Studioに従来搭載されているプログラムによって予測することが可能です。これらのツールにより、研究者はタンパク質の安定性や物性、リガンドとの相互作用の強さを総合的に予測しながら、変異導入箇所の優先順位付けを行うことができます。
また、CDRの疎水性を予測することによる、開発候補抗体の優先順位付けも可能です。この他、DS Protein Aggregationは、抗体以外のタンパク質-タンパク質相互作用部位の特定にも有効にお使いいただけます。
お気軽にお問い合わせください
 電話でお問い合わせ
電話でお問い合わせ
- 東京(担当:SATグループ)
- 03-3520-3082
受付時間 9:00-17:30(土・日・祝除く)

 製造業向けITソリューション
製造業向けITソリューション  建設業務改善ソリューション
建設業務改善ソリューション  ビル管理業務支援 DK-CONNECT BM
ビル管理業務支援 DK-CONNECT BM FILDER CeeD TOP
FILDER CeeD TOP  FILDER CeeD 電気 TOP
FILDER CeeD 電気 TOP  Rebro D TOP
Rebro D TOP  データ・サイエンス・ソリューション Pipeline Pilot
データ・サイエンス・ソリューション Pipeline Pilot 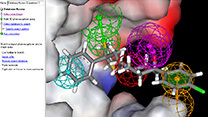 ライフサイエンス向けソフト Discovery Studio
ライフサイエンス向けソフト Discovery Studio 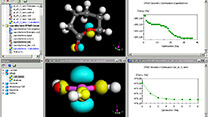 マテリアルサイエンス向けソフト Materials Studio
マテリアルサイエンス向けソフト Materials Studio  電子実験ノート
電子実験ノート 総合3DCG 制作ソフト Maya
総合3DCG 制作ソフト Maya  総合3DCG 制作ソフト 3ds Max
総合3DCG 制作ソフト 3ds Max  総合3DCG 制作ソフト MODO
総合3DCG 制作ソフト MODO  アニメーション制作ソフト Toon Boom
アニメーション制作ソフト Toon Boom